| はじめに |
 | 遺伝子を操作して目的とする遺伝子を破壊したり、別の遺伝子を挿入する「ゲノム編集」技術は激しい勢いで開発がすすみ、医療分野や従来の遺伝子組換えにとって代わる日が目の前に来ている。その一方で、何十億年もかけて進化した生物の遺伝子を自在に改変出来る技術を手にした人間が、地球の未来にも影響を与えうる立場にいることを私たちは改めて自覚し、生命を操作するゲノム編集の是非について自らの責任を問わなければならない。私たちの社会が新しい技術を受け入れるか否かを判断するに際し、その技術の何が問題かを明らかにし、その解決に取り組む必要がある。ここでは、ゲノム編集の技術的課題の一つである「オフターゲット」について最新の知見をもとに考察する。
|
| 1)オフターゲットとは |
 |
改変の標的となる遺伝子は、通常1個(未受精卵など半数体の場合)又は2個(受精卵又は体細胞など2倍体の場合)である。その為に、人工 DNA 分解酵素の ZFN や TALEN、細菌由来の CRISPR/Cas9 等を作る遺伝子を、通常はベクターと呼ばれる自己増殖機能を壊したウイルスやプラスミドの DNA の中に埋め込み、標的細胞に感染させて細胞内で DNA 分解酵素を作らせ、それによって細胞の遺伝子 DNA を切除(ノックアウト)したり、新たな DNA を挿入(ノックイン)する。その際に、標的遺伝子以外の他の遺伝子にも同様の影響をもたらす。これをオフターゲト作用という。最近はゲノム編集酵素をベクターを使わずに生分解性のナノパーティクルに封入し、細胞に直接打ち込む技術(エレクトロポレーション)も開発されている。この場合もオフターゲットは起こる。
ゲノム編集の目的からすればこうした事象は起こってはならないが、様々な要因によりオフターゲットは避けられない。その結果宿主細胞にとって意図しない影響が生ずるがそれをあらかじめ予測する事は出来ない。 |
2)オフターゲットの原因(その1)
DNA分解酵素による標的遺伝子DNAの塩基配列の認識エラー |
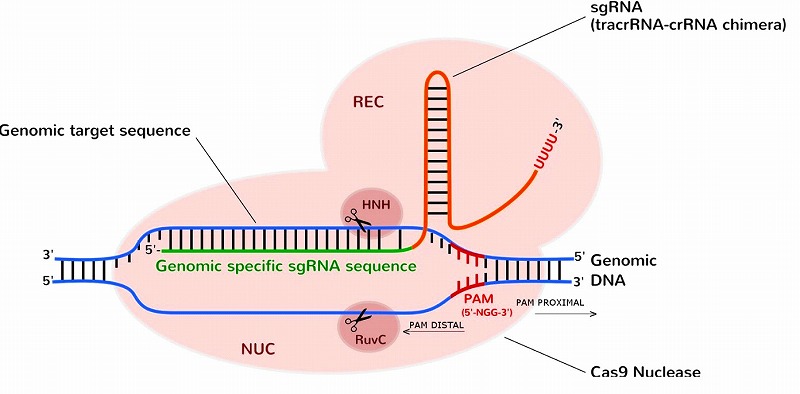
| DNA の2重らせんを作っている相補的な塩基配列どうしでは、お互いの認識エラー(ミスマッチ)は殆ど起こらないが、ゲノム編集の場合、標的 DNA の塩基配列を認識するのは、ZFN と TALEN では DNA 分解酵素(蛋白質)自身であり、 CRISPR/Cas9 の場合はガイド RNA(gRNA)と呼ばれる RNA である。これまでの報告では、ZFN と TALEN よりも、最近よく使われるCas9 の方がオフターゲットの起こる確率が高い。Cas9 の場合、標的 DNA の塩基配列を認識する gRNA の塩基配列は約20個だが、相手の DNA の塩基配列が数個違っても結合し、Cas9 酵素が切断してしまう。その結果 Cas9 によるオフターゲットは非常に多く起こる(1)。その大きな理由の一つは、標的 DNA を認識する gRNA の長さは20塩基と言われているが、実際には標的 DNA の認識には20塩基の gRNA の配列の中、Cas9 酵素(蛋白質)がまず認識する標的 DNA 中の PAM と呼ばれる通常3個の塩基配列(NGG:図参照)とそれに隣接する1個〜5個の塩基配列(seed 配列と呼ばれる)が大きな役割をしており、PAM 配列から遠いほど、ミスマッチがあっても Cas9 による DNA の切断は起こってしまう(2)。即ち20塩基全てが標的 DNA 配列との認識の精度に関わっているわけではない(1)。また膨大な数の塩基対からなるゲノム DNA には数塩基程度が共通な遺伝子は数多くあり、その結果 gRNA が間違った標的に Cas9 酵素を結合させるリスクは大きい。実際、ヒト細胞の Cas9 を使ったゲノム編集でRNA-DNAの配列のミスマッチによる大量のオフターゲットが観察されている(3)。 |
3)オフターゲットの原因(その2)
1個の遺伝子から複数の蛋白質を作るスプライシングが原因のオフターゲット |
|
真核生物の遺伝子は蛋白質のアミノ酸配列に反映される配列(エクソン)と反映されない配列(イントロン)から構成されている。例えばヒトの筋肉細胞を作るジストロフィン遺伝子は250万個の塩基対、79個のエクソンから出来ている。実際に蛋白質を作るメッセンジャーRNA(mRNA)は DNA の全ての塩基配列をコピーした pre-mRNA をスプライシング酵素が必要なエキソン同士を選択し、つなぎ合わせて作られる。つなぎ合わせるエクソンが違えば異なる蛋白質が造られる。即ち、1個の遺伝子から複数の蛋白質が造られるのである。ゲノム編集で必要なエクソンを切除したり、そこに外来 DNA を挿入した場合、複数の蛋白質に共通のエクソンを編集すれば、結果的に複数の蛋白質を編集したことになりオフターゲットが生ずる。事前に標的遺伝子の該当エクソンが1個の蛋白質にしか使われていない事が分かれば問題ないが、現在、多くの生物で DNA の全構造は解明されつつあるものの、蛋白質の数や種類は依然として不明である。ヒトゲノムの場合、全塩基対は31億個、遺伝子の数は 22000 個だが、それから作られる蛋白質の種類と数は数万〜数十万個といわれ、未だに正確には解っていない。特定の遺伝子の1個のエクソンを破壊しても、複数の蛋白質に影響を及ぼす可能性は否定できないのである。
|
4)オフターゲットの原因(その3)
ゲノム編集に使う DNA 分解酵素や gRNA の濃度 |
|
ゲノム編集を行う細胞1個(遺伝子数は半数体の場合は1個、2倍体の場合は2個)に対して、細胞内に投入される ZFN や TALEN, Cas9(+gRNA)の過剰な濃度もオフターゲトの原因である。Cas9 と gRNA の濃度と標的細胞の数(標的遺伝子の数)によるオフターゲットの頻度を調べた実験がある(2)。それによれば、細胞1個当り 1.8×10(−11乗)から7.1×10(−10乗)nm(ナノモル:Cas9-gRNAプラスミドの重量で 10ng 〜400ng:ナノグラム)の濃度(量)で変化させると、濃度が高くなるにつれ劇的にオフターゲットが増加する。これを Cas9-gRNA の分子の数に直すと以下のようになる。
Cas9-gRNA の分子数:
1.8×10(−11乗)nm= 1.8×10(−11乗)×6×10(14乗)=1.08×10(4乗)個。
7.1×10(−10乗)nm=7.1×10(−10乗)×6×10(14乗)=4.26×10(5乗)個。
但し、分子数=1nm(ナノモル)=1×10(−9乗)×アボガドロ数
=1×10(−9乗)×6×10(23乗)=6×10(14乗)個
即ち、ゲノム編集には一個の細胞に含まれる標的遺伝子(n=1~2)に対して、10,000個〜100,000個もの Cas9-gRNA を投入しているのである。他のゲノム編集実験でもこの量比は似たり寄ったりである。実験によっては細胞一個当たり10(8〜10乗)個の(gRNA や Cas9 酵素を持つ)ベクターを感染させている。100,000 本もの矢を放てば的に当たる矢があるのは当然として、標的から外れた場所に当たる矢(オフターゲット)が生ずるのは当然である。勿論、投入した全てのベクターやナノパーティクルが細胞内に入るわけではないが、標的一個に対する多数の DNA 分解酵素の存在は大きなリスクの原因である。ベクター(プラスミド)濃度の増加によるゲノム編集効率(オンターゲット)向上とその結果によるオフターゲット増加は大きなジレンマである。ナノパーティクルによる細胞注入は1回きりだが、ウイルスやプラスミド等のベクターで打ち込む場合は、細胞内で Cas9-gRNA が常時合成・増殖され、標的遺伝子との濃度差はさらに大きくなる。
|
5)ZNF 及び TALEN によるオフターゲット |
| 以上、Cas9 酵素によるオフターゲットについて述べたが、同様の事は、ジンクフィンガー(ZNF)やターレン(TALEN) DNA 分解酵素でも起こることが確認されている(4,5)。 |
6)オフターゲットの検出の必要性 |
| 上記のようにゲノム編集に伴うオフターゲットの発生は大きな問題である。従ってゲノム編集でオフターゲットが生じたか否かの検出を厳密に行わなければならない。厚労省内には、生物の全遺伝子構造が分かっていないものも多くオフターゲットの検出を義務化するのは困難との意見もあるが、この考えは本末転倒である。 |
 |
| 文献 |
| (1) |
Xiao-Hui Zhang etal.: Off-target Effects inCRISPR/Cas9-mediated Genome Engineering.
Molecular Therapy-Nucleic Acid (2015) 4e 264. |
| (2) | Patrick D.Hsu etal.: DNA targeting specificity of RNA-guided Cas9 nucleases.: Nature Biotechnology.(2013) vol.31 p827-832. |
| (3) | Yanfang Fu etal. : High frequency off-target mutagenesis induced by CRISPR/Cas9 nucleases in human cells. : Nat.Biotechnology. (2013) September;31(9):822-826. |
| (4) | Thomas J Cradick et.al.: ZFN-Site searches genomes for Zinc fnger nuclease target sites and off-target sites.: BMC Bioinformatics(2011),vol.12. 152 |
| (5) | John P. Gullinger et.al.: Broad Specificity Profiling of TALENs Results in Engineered Nucleases With Inmroved DNA Cleavage Specificity.: Nature Methods.(2014) April. Vol.11(4):429-435. |